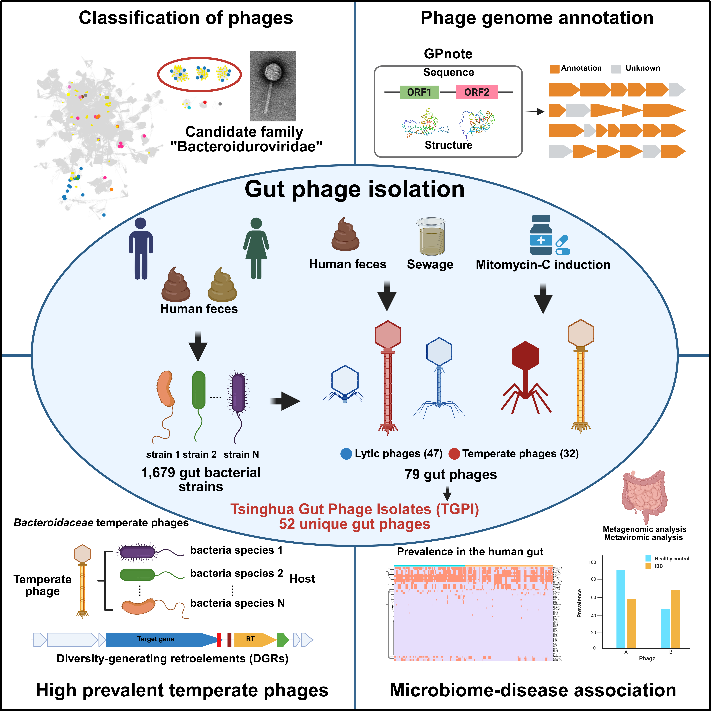
2025年12月9日,清华大学基础医学院向烨团队在期刊Gut Microbes上发表题为“Extensive cultivation of human gut phages revealing undescribed Bacteroidaceae phages”的研究论文。该研究通过大规模培养与分离,成功获得一系列裂解型及温和型肠道噬菌体并构建了相应的噬菌体资源库。进一步团队测定了所分离噬菌体的基因组序列,开发了基于结构的噬菌体基因功能注释工具,实现了对所分离噬菌体57.5%基因的功能注释,并基于此进行聚类分析发现一个全新的拟杆菌噬菌体家族,为深入解析肠道微生物组的调控机制提供了重要资源与工具。

长期以来,肠道噬菌体研究受限于其“难分离、难培养”的特点,已报道的研究多集中于裂解型噬菌体,而对温和型噬菌体的认知仍存在显著空白。为突破这一局限,研究团队首先从37名健康捐赠者的粪便样本中,采用20种不同的培养条件,成功分离出1679株肠道细菌,涵盖86个物种。以此为基础,团队进一步从粪便及污水样本中分离得到79株可感染肠道细菌的噬菌体,最终筛选出52个非冗余噬菌体,构建了“清华肠道噬菌体分离库(Tsinghua Gut Phage Isolates,TGPI)”。
值得注意的是,研究发现部分拟杆菌温和型噬菌体相较于已知裂解型噬菌体,表现出更高的基因组多样性与人群流行率。这些温和型噬菌体可在不同拟杆菌科物种的基因组中被检测到,提示其可能具有广泛的宿主范围。基因组分析进一步揭示,多数具有潜在广宿主特性的温和型噬菌体携带“多样性生成逆转录元件(DGRs)”,该元件可通过靶向特定基因变异帮助噬菌体适应不同宿主。上述发现为理解噬菌体与细菌的协同进化提供了新线索,同时也提示温和型噬菌体可能在肠道微生态中扮演比以往认知更为重要的角色。
针对肠道噬菌体基因序列相似度低、功能注释困难的研究瓶颈,团队开发了新型注释工具GPnote(https://github.com/MaoSihong/GPnote)。该工具创新性整合了“序列比对”与“结构预测”双策略,大幅提升了噬菌体基因组的注释率,尤其对噬菌体头部、尾部等关键结构蛋白的注释率达到90%,为高效解析噬菌体功能基因提供了可靠手段。借助GPnote的精准注释,团队发现了一组具有非收缩长尾形态,以拟杆菌科细菌为宿主的裂解型噬菌体,与已知噬菌体家族的平均氨基酸相似度(AAI)低于5%。进化分析表明,这类噬菌体形成一个独立的进化分支,在演化早期便与其他噬菌体分化。基于此,团队提出了一个全新的噬菌体候选家族,暂命名为“Bacteroiduroviridae”,并从公共数据库中挖掘出196条同源序列,证实该家族在自然界中广泛存在,为噬菌体分类学研究增添了潜在新成员。
该研究还初步揭示了肠道噬菌体与疾病的潜在关联。从健康人粪便中分离的温和型噬菌体BX-TP1(宿主为解木聚糖拟杆菌),在炎症性肠病(IBD)患者样本中的流行率显著低于健康人群;而感染活泼瘤胃球菌的温和型噬菌体RG-TP1,在IBD患者中的流行率则明显升高。这些发现为后续探索噬菌体在肠道相关疾病发生发展中的作用提供了重要方向。
该研究不仅为肠道噬菌体研究提供了宝贵的生物资源与高效的分析工具,也进一步拓展了对肠道微生态复杂性的认知。未来,基于这一资源库的深入研究,将有助于系统揭示肠道微生物组的多样性、功能及其与宿主的互作机制,为相关疾病的预防与治疗提供新思路。
清华大学基础医学院教授,清华北大生命联合中心、北京生物结构前沿研究中心、山西医科大学-清华大学前沿医学研究中心研究员向烨与已出站博后袁玉玉博士(现清华大学基础医学院微生物平台主管)为该论文的通讯作者。清华大学基础医学院博士生刘哲、杨耀宇、毛思闳为共同第一作者。英才计划高中生王兆琪(现纽约大学本科在读)、博士生朱启衡参与了部分研究工作。清华大学基础医学院梁冠翔副教授为本研究提供了部分数据集并对手稿提出了修改意见。本研究获国家重点研发计划、国家自然科学基金、山西医科大学-清华大学前沿医学协同创新中心、清华-北大生命科学联合中心等项目经费的支持。
原文链接https://doi.org/10.1080/19490976.2025.2597614