组蛋白修饰是表观遗传调控重要机制,在复制、转录和修复等以染色质为模板的细胞过程中发挥着至关重要的作用。参与组蛋白识别的因子被形象地称作组蛋白“阅读器”(reader)。自1999年美国西奈山医学院的周明明(Ming-Ming Zhou)实验室发现第一类组蛋白阅读器—溴域(Bromodomain)以来,发现新型“组蛋白-阅读器”识别对并探究其生物功能成为表观遗传学领域的一个研究热点。
2021年9月7日,清华大学医学院李海涛课题组于《Nucleic Acids Research》发表题为Molecular basis for bipartite recognition of histone H3 by the PZP domain of PHF14(PHF14的PZP结构域二分法识别组蛋白H3的分子基础)的研究论文,报道了PHF14(Plant homeodomain finger protein 14)蛋白作为一种新鉴定的组蛋白H3阅读器,通过其PZP(PHD1-Znk-PHD2)结构域,“二分法”识别组蛋白H3氨基端柔性尾巴1-34的独特机制。该工作揭示了PZP结构域家族新的组蛋白结合活性,并在分子水平上阐释了PHF14的生物学功能和作用机理。

PHF14全长含有3-4个PHD锌指结构域。作为一类表观调控因子,PHF14在胚胎发育,癌症发生和组织稳态维持等过程中均发挥着重要作用。例如,纯合敲除PHF14的小鼠在出生几个小时候就会因为呼吸衰歇而死亡,其肺部组织纤维化,其他组织器官组织发育异常1,2。PHF14负调控PDGFRα、p14AR、p16INK4a和p15INK4b等表达,进而调控细胞周期和癌症发生过程1-8。尽管PHF14的生物学功能十分重要,但其分子功能,尤其是其PHD锌指结构域的组蛋白识别活力仍不清晰。
本研究通过结构生物学、生物化学以及氢氘交换质谱等手段,首次发现PHF14的PZP结构域是一个超长组蛋白H3氨基端尾巴的高效阅读器,结合常数约为200纳摩尔。相比之下,位于PHF14的第三个和第四个PHD锌指则不具备类似识别能力。PHF14PZP采用两个截然不同表面实现对非修饰组蛋白H3(1-34)的分段识别,即采用“β1-insertion loop”表面去识别组蛋白H3的氨基端H3(1-9);而用“α-ZP”面去识别组蛋白H3中间段H3(14-34)。有意思的是,研究发现PHF14PZP对组蛋白H3的氨基端和中间段的识别既可同时发生,又可相对独立进行,打破其中一段的识别会导致结合力下降10倍至2微摩尔左右,相比于其它组蛋白识别事件,这仍是一种较强的结合。这种既同时发生又相对独立的分段识别模式即“二分法”识别。
令人惊讶的是,该研究发现了一种全新的PHD 锌指识别H3的模式。目前已知PHD锌指结构域识别配体的模式一共有四种,即β1面识别、β1-N面识别、β2面识别和α1面识别。本研究中PHF14PHD1识别H3的模式不属于以上任何一种,是一种崭新识别模式(图1)。在经典的β1面识别中,组蛋白H3与β1反向平行,K4顺式伸向β2方向,而在PHF14PZP识别H3中,PHF14PZP特有的一个柔性插入环(insertion loop)了序列主导H3K4的识别。晶体结构研究揭示H3K4反向插向该loop区,而非经典的β1-β2核心口袋。值得注意的是,这个插入环在其它拥有类似组蛋白H3识别能力的旁系同源蛋白,诸如AF10、BRPF1、BHC80、BPTF和ING2等中均不存在,表明PHF14采用了一种特有的PHD锌指分子设计来实现组蛋白识别功能。利用序列和结构上并不保守的一段插入序列来“获得”组蛋白识别能力,打破了人们对PHD锌指识别H3K4结构和机制保守性的认识。本工作也是自2006年首次发现PHD锌指具备组蛋白H3K4识别功能以来,第一次揭示出PHD识别H3K4的趋异化结构设计。这一方面为根据同源结构域关键残基保守性来预测潜在阅读器组蛋白识别能力的策略提供了反例,另一方面提示了更多新型阅读器有待被发现的可能,同时充分表明实验科学的必要性和重要性。
另一个值得关注的地方是,PHF14的直系同源序列比对显示,该插入环序列在从鱼至人的物种中高度保守,但在果蝇和线虫中并不保守,表明PHF14在进化至脊椎动物后才获取了组蛋白阅读器活性。该现象让人联想到CHD1蛋白,人源CHD1的double Chromo结构域能够识别H3K4me3,而同源的酵母CHD1则没有识别H3K4me3活性,这可能与单细胞酵母不需要CHD1-H3K4me3识别来保证基因表达复杂度有关。对于PHF14的分子设计而言,无论是横向旁系还是纵向直系同源进化,只有亟需组蛋白识别功能的PHF14家族才“后天获得”了组蛋白识别功能,提示组蛋白阅读器在功能需求和结构支撑上存在协同进化。如此“精妙而人为”的分子进化设计与抗体利用可变区快速进化出抗原表位识别能力的过程相类似。分子识别,尤其是修饰介导的分子识别,所能够带来的丰富与多样永远值得人们期待!
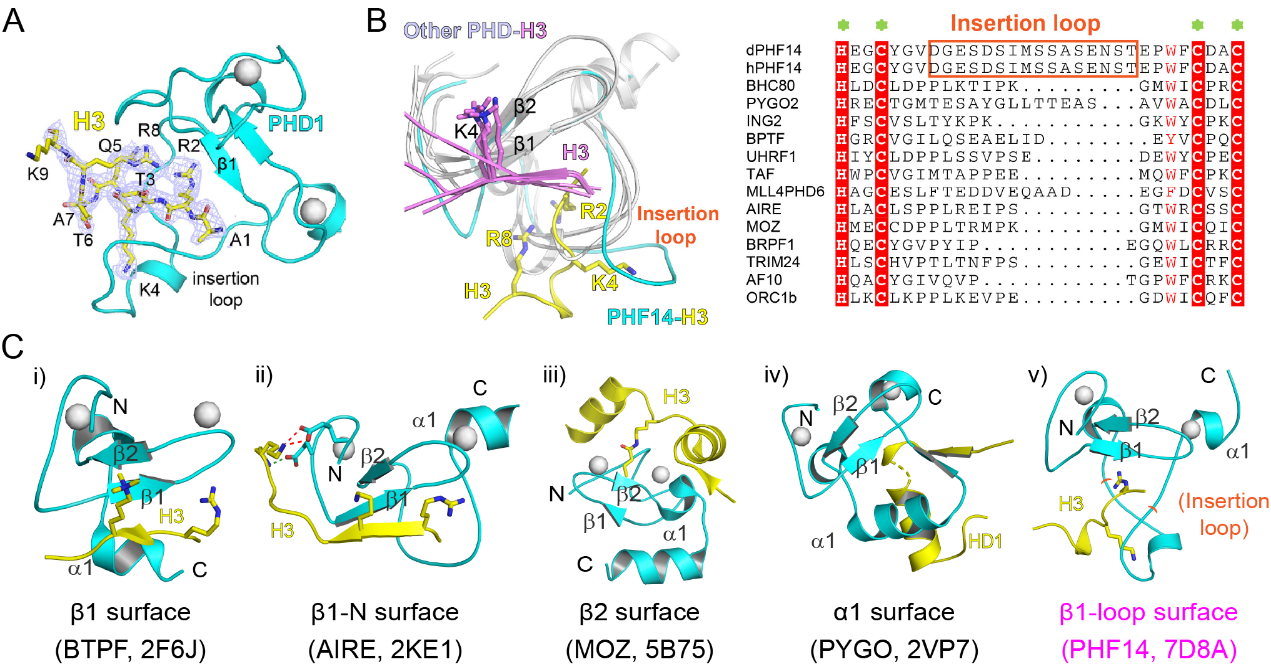
图1. PHF14PZP由独特insertion loop 主导对H3氨基端识别
进一步的修饰交叉会话研究发现,PHF14对H3R2甲基化、T3磷酸化、K4甲基化、R8甲基化和K23乙酰化等活跃转录相关的修饰敏感,但可以容忍K9三甲基化和K27三甲基化修饰。已有研究表明PHF14是PDGFRα,p14ARF,p15INK4b和p16INK4a的负调控因子,本研究实验结果暗示PHF14具备抑制或者维持这些基因的非活跃状态的功能。PHF14作为一种非修饰的“基”态H3(1–34)阅读器,在维持靶基因非活跃状态的同时,或可因为活跃转录相关修饰的建立而实现去抑制,从而以一种层次性去抑制的策略,响应上游信号,促进靶基因从“非活跃“到”活跃“状态的切换,实现基因表达的命运决定。因此,本工作的研究成果为后续围绕PHF14的功能研究提供了重要机理支撑和理论指导。
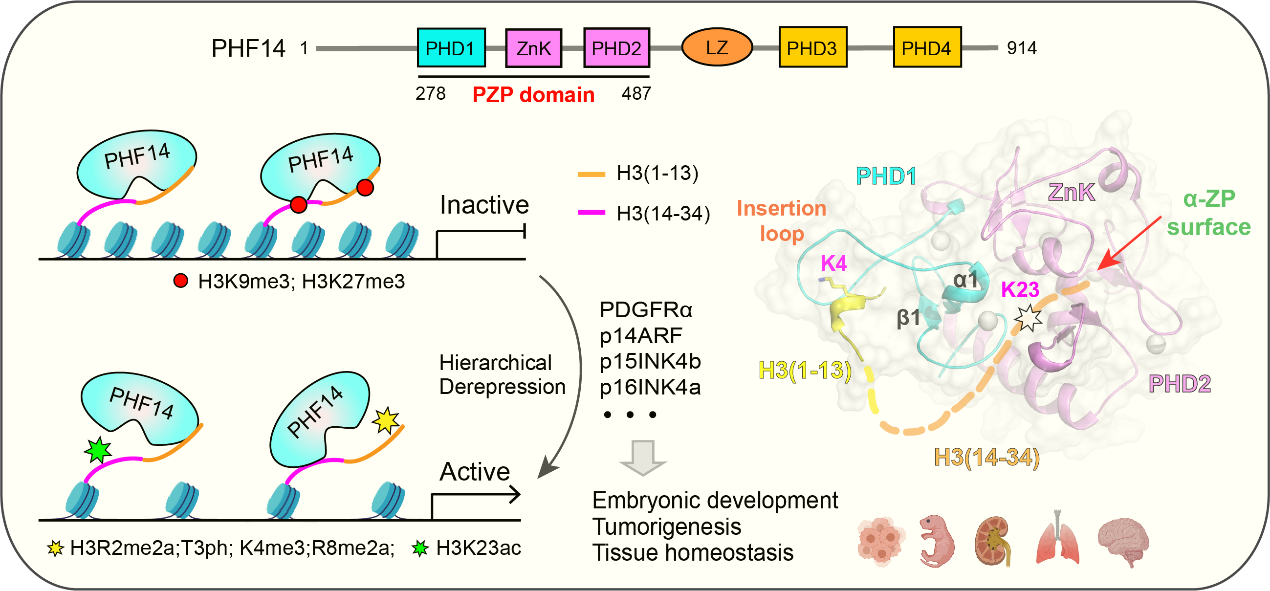
图2. PHF14对H3识别的分子机制及分子功能阐释
本研究成果在清华大学医学院完成。李海涛教授为通讯作者,郑双平博士为第一作者,毕于聪硕士以及陈海宁同学对本研究作出了重要贡献。本工作还得到了生命学院龚波博士和贾顺姬老师的指导和支持。同时,本工作也得到了国家蛋白研究中心(上海),清华大学蛋白质化学与组学平台质谱平台以及清华大学X-ray晶体学平台的大力支持与帮助。
原文链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab670/6345469

扫描上方二维码直达原文链接
参考文献:
1. Kitagawa, M. et al. Phf14, a novel regulator of mesenchyme growth via platelet-derived growth factor (PDGF) receptor-alpha. J Biol Chem 287, 27983-27996, doi:10.1074/jbc.M112.350074 (2012).
2. Huang, Q. et al. Depletion of PHF14, a novel histone-binding protein gene, causes neonatal lethality in mice due to respiratory failure. Acta biochimica et biophysica Sinica 45, 622-633, doi:10.1093/abbs/gmt055 (2013).
3. Zhao, Y. et al. PHF14 Promotes Cell Proliferation and Migration through the AKT and ERK1/2 Pathways in Gastric Cancer Cells. Biomed Res Int 2020, 6507510, doi:10.1155/2020/6507510 (2020).
4. Wu, S. et al. Silencing expression of PHF14 in glioblastoma promotes apoptosis, mitigates proliferation and invasiveness via Wnt signal pathway. Cancer Cell Int 19, 314, doi:10.1186/s12935-019-1040-6 (2019).
5. Miao, L., Liu, H. Y., Zhou, C. & He, X. LINC00612 enhances the proliferation and invasion ability of bladder cancer cells as ceRNA by sponging miR-590 to elevate expression of PHF14. J Exp Clin Cancer Res 38, 143, doi:10.1186/s13046-019-1149-4 (2019).
6. Jung Eun Park, S. W. T., Guo Xue, Christina AssisiMaqueda1, Gallart Palau Xavier Ramon1, Jee Keem Low2, Oi Lian Kon3 Tay4, James P. Tam1 and Siu Kwan Sze. Pulsed SILAC-based proteomic analysis unveils hypoxia- and serum starvation-induced de novo protein synthesis with PHD finger protein 14 (PHF14) as a hypoxia sensitive epigenetic regulator in cell cycle progression. Oncotarget 10, 2136-2150 (2019).
7. Zhang, L. et al. A novel PHD-finger protein 14/KIF4A complex overexpressed in lung cancer is involved in cell mitosis regulation and tumorigenesis. Oncotarget 8, 19684-19698, doi:10.18632/oncotarget.14962 (2017).
8. Akazawa, T. et al. Aberrant expression of the PHF14 gene in biliary tract cancer cells. Oncology letters 5, 1849-1853, doi:10.3892/ol.2013.1278 (2013).