2019年9月17日,清华大学医学院长聘副教授,北京结构生物学高精尖创新中心研究员,北京生物结构前沿研究中心研究员向烨团队在《Cell Research》在线发表题为《Structure of the African swine fever virus major capsid protein p72(非洲猪瘟病毒衣壳蛋白p72的结构)》的学术论文。该研究采用冷冻电镜单颗粒三维重构的方法首次解析了非洲猪瘟病毒衣壳蛋白p72的高分辨率结构,揭示了非洲猪瘟病毒衣壳可能的组装机制,为非洲猪瘟病毒亚单位疫苗的开发打下坚实基础。
非洲猪瘟病毒起源于非洲,家猪感染致死率近100%,对家猪饲养产业乃至民生经济都有重大的威胁。非洲猪瘟病毒在过去的几十年时间从非洲迅速传播到了欧洲,南美洲以及亚洲的一些地区。2018年8月我国东北地区发现非洲猪瘟病病毒感染,很快病毒传播到全国大部分地区,造成超过千亿人民币的经济损失。目前紧迫需要研发有效的疫苗和防疫手段控制及预防非洲猪瘟病毒。应国家之急需,由清华大学北京结构生物学高精尖创新中心及北京生物结构前沿研究中心牵头,联合北京大学,中国农业科学院哈尔滨兽医研究所,上海巴斯德所等单位十几个研究组于2019年4月开始非洲猪瘟病毒攻关研究。
非洲猪瘟病毒是一种双链DNA病毒,拥有复杂的结构,它的基因组由两层衣壳和两层外膜保护,衣壳主要由病毒编码蛋白p72构成,据以往试验数据估计p72占病毒颗粒总重33%左右,同时p72是被病毒感染猪血液中能检测到的主要抗原。在研究p72蛋白初期,向烨研究组发现单独重组表达p72蛋白不能得到有正确折叠构象的p72蛋白,只有在p72和非洲猪瘟编码的另一个辅助蛋白B602L共同表达的情况下才可以得到正确折叠构象的p72蛋白。以此为前提,研究组使用冷冻电镜单颗粒三维重构的方法确定了p72蛋白2.67 Å 的高分辨率三维结构(图1)。结果显示三个p72分子形成了稳定的三聚体形式。每个p72包含两个串联的卷筒蛋糕样结构域,每个结构域由8个β折叠片组成,这种结构域在大正二十面体病毒衣壳蛋白结构中很常见。三聚体p72的六个卷筒蛋糕样结构一起形成了一个伪六聚体的底部锚定在病毒的内膜上。三聚体p72折叠片之间的插入片段一起形成了一个类似于螺旋桨一样的顶部结构伸向病毒外部,极有可能是细胞表面受体结合区域。
此前基于非洲猪瘟病毒p72蛋白的亚单位疫苗研究已有很多尝试,但没有获得良好结果。分析前期相关研究失败原因很可能是没有使用正确折叠的p72蛋白。正确折叠p72蛋白的获取以及其高分辨率结构的确定有助于我们开发靶向病毒衣壳组装的药物以及亚单位疫苗。目前p72亚单位疫苗的研发及评估正由清华大学与中国农业科学院哈尔滨兽医研究所联合紧密进行中。
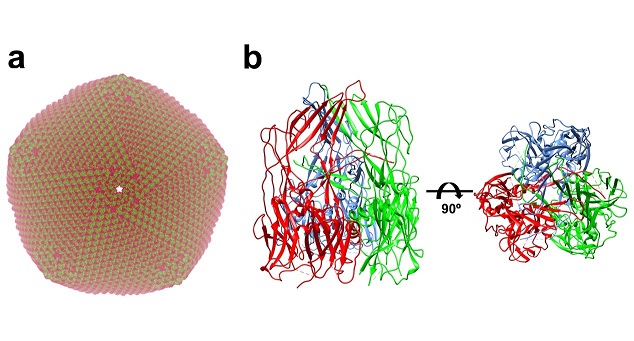
图1: 非洲猪瘟病毒结构。a. 依据非洲猪瘟病毒衣壳蛋白p72结构模拟病毒组装产生的非洲猪瘟病毒衣壳结构。b. 非洲猪瘟病毒衣壳蛋白p72三聚体冷冻电镜结构。
清华大学医学院向烨副教授为本文通讯作者。清华大学生命联合中心2014级博士生刘琦为本文第一作者,向烨实验室的2015级博士生马丙婷,清华大学药学院钱年超和张帆,清华大学生命学院校级冷冻电镜平台主任雷建林教授参与了该工作。该研究得到了国家蛋白质科学研究(北京)清华大学基地冷冻电镜平台的大力支持,且得到了国家自然科学基金,科技部重大研发计划,北京结构生物学高精尖创新中心以及北京生物结构前沿研究中心的大力支持。
文章链接:https://www.nature.com/articles/s41422-019-0232-x